Online-Artikel: Datenanalyse und künstliche Intelligenz in Python
über
Von Angelo Cardellicchio
Begriffe wie Big Data und Künstliche Intelligenz sind inzwischen zu einem festen Bestandteil der Alltagssprache geworden. Dies ist vor allem auf zwei Faktoren zurückzuführen: Zum einen hat die zunehmende und weit verbreitete Verbreitung von Datenerfassungssystemen die Schaffung praktisch endloser Repositories des Wissens ermöglicht, zum anderen hat die Zunahme der Rechenkapazitäten, die auch auf den weit verbreiteten Einsatz von GPUs [1] zurückzuführen ist, es ermöglicht, Probleme zu bewältigen, deren Lösung einst unmöglich war.
Nehmen wir ein Beispiel, das wir in diesem Artikel als Anwendungsszenario verwenden werden. Stellen wir uns vor, wir müssten eine ganze Produktionskette überwachen (unabhängig vom Endprodukt, das in diesem Zusammenhang keine Rolle spielt). Wir haben die Möglichkeit, Daten aus verschiedenen Quellen zu erfassen: Wir könnten zum Beispiel Sensoren entlang der gesamten Produktionslinie einsetzen oder Kontextinformationen verwenden, die sich auf das Alter und den Typ jeder Maschine beziehen. Dieser Datensatz (Dataset) kann für verschiedene Zwecke verwendet werden: Die vorausschauende Wartung ermöglicht uns beispielsweise, den Beginn anormaler Situationen zu bewerten und vorherzusagen, um einen Austausch oder eine Reparatur vor dem Ausfall zu planen, was zu Einsparungen und erhöhter Produktivität führt. Darüber hinaus ermöglicht die Kenntnis der Datenhistorie eine Korrelation der von jedem Sensor gemessenen Größen, wodurch mögliche Ursache-Wirkungs-Beziehungen aufgezeigt werden. Wenn beispielsweise auf einen plötzlichen Anstieg der Temperatur und der Luftfeuchtigkeit in einem Raum ein Rückgang der Anzahl der gefertigten Teile folgt, kann es erforderlich sein, die Klimaanlage des Raumes zu verbessern, um konstante klimatische Bedingungen aufrechtzuerhalten.
Die Implementierung eines solchen Systems ist sicherlich nicht für jeden erreichbar, wird aber durch die von der Open-Source-Community zur Verfügung gestellten Werkzeuge vereinfacht. Für den Anfang brauchen Sie also nur einen PC (oder alternativ auch unseren treuen Raspberry Pi, wenn die zu verarbeitende Datenmenge nicht zu groß ist), Kenntnisse in Python (die Sie durch Tutorials [2] vertiefen können) und natürlich die Tools, das heißt, die Konzepte und grundlegenden Werkzeuge. Lassen Sie uns diese gemeinsam entdecken!
Die Tools
Unnötig zu sagen, dass wir zunächst in der Lage sein müssen, Programme zu erstellen, die in der Sprache Python geschrieben sind. Dazu müssen wir den Interpreter installieren, der auf der offiziellen Website von Python [3] zu finden ist. Im weiteren Verlauf dieses Artikels gehen wir davon aus, dass Sie Python bereits installiert und zu den Umgebungsvariablen des Systems hinzugefügt haben.
Die virtuelle Umgebung
Sobald die Einrichtung von Python abgeschlossen ist, wird es an der Zeit sein, unsere virtuelle Umgebung einzurichten, eine Art „Container“, der vom Rest unseres Systems abgeschottet ist und in dem wir die von uns verwendeten Bibliotheken installieren werden. Der Grund, warum eine virtuelle Umgebung einer globalen Installation von Bibliotheken vorzuziehen ist, hängt vor allem mit der raschen Entwicklung in der Python-Welt zusammen. Sehr oft gibt es erhebliche Unterschiede zwischen den Releases des Interpreters, was dazu führt, dass Bibliotheken (und damit Programme) inkompatibel werden. Eine deterministische Umgebung zu haben, von der wir die Versionen jeder einzelnen installierten Bibliothek im Detail kennen, stellt eine Art „Garantie“ für das Funktionieren unserer Programme dar: Es reicht tatsächlich aus, die Konfiguration der virtuellen Umgebung exakt nachzubilden.
Zur Verwaltung unserer virtuellen Umgebungen verwenden wir ein Paket namens virtualenvwwrapper, das von der Shell mit dem Paketmanager pip installiert wird:
$ pip install virtualenvwrapper
Sobald die Installation abgeschlossen ist, erstellen wir eine neue virtuelle Umgebung:
$ mkvirtualenv ml-python
ml-python ist dabei der Name der im Beispielszenario gewählten virtuellen Umgebung. Natürlich kann der Name auch anders sein und Sie können den Namen wählen, der Ihnen am besten gefällt. Die virtuelle Umgebung wird nun aktiviert:
$ workon ml-python
Wir sind nun zur Installation der für den Rest des Artikels notwendigen Bibliotheken bereit.
Die Bibliotheken
Die Bibliotheken, die wir beschreiben und verwenden werden, gehören zu den fünf am häufigsten für die Datenanalyse in Python eingesetzten.
Die erste und vielleicht berühmteste ist Numpy, eine Art Portierung von MATLAB in Python. Numpy ist eine Bibliothek vor allem für algebraische und Matrix-Berechnungen. Wer gewöhnlich MATLAB verwendet, dürfte viele Ähnlichkeiten finden, sowohl in Bezug auf die Syntax als auch auf die Optimierung. Die Verwendung algebraischer Berechnungen in Numpy ist effizienter als verschachtelte Zyklen, gerade mit MATLAB (in dem Tutorial [4] erfahren Sie mehr darüber). Der Datentyp, der einer Numpy-Berechnung zugrunde liegt, ist das Array, nicht zu verwechseln mit dem bei Computerberechnungen üblichen Vektor, sondern im algebraischen und geometrischen Sinne als Matrix zu verstehen. Da die Datenanalyse auf algebraischen und Matrix-Operationen basiert, ist Numpy auch die „Basis“ für zwei der am häufigsten verwendeten Frameworks in diesem Bereich, nämlich Scikit-Learn (dazu gleich mehr) und TensorFlow.
Eine natürliche Ergänzung zu Numpy ist Pandas, eine Bibliothek, die zur Verwaltung und zum Lesen von Daten aus verschiedenartigen Quellen wie Excel-Tabellen, CSV-Dateien oder sogar JSON- und SQL-Datenbanken geeignet ist. Pandas ist extrem flexibel und leistungsstark und ermöglicht es Ihnen, Daten in Strukturen, so genannten Dataframes, zu organisieren, die nach Belieben manipuliert und problemlos direkt in Numpy-Arrays übernommen werden können.
Die dritte Bibliothek, die wir verwenden werden, ist Scikit-Learn. Scikit-Learn, das aus einem akademischen Projekt hervorgegangen ist, ist ein Framework, das die meisten der heute verwendeten Algorithmen des maschinellen Lernens implementiert und eine gemeinsame Schnittstelle bietet. Dieses Konzept ist genau das der objektorientierten Programmierung: Es ist möglich, durch die Methode fit_transform praktisch jeden von Scikit-Learn angebotenen Algorithmus zu verwenden, an den mindestens zwei Parameter übergeben werden, das bedeutet, die zu analysierenden Daten und die mit ihnen verbundenen Labels.
Die letzten beiden verwendeten Bibliotheken sind Matplotlib und Jupyter. Die erste ist zusammen mit dem Addon Seaborn notwendig, um die Ergebnisse unserer Experimente in grafischer Form zu visualisieren; die zweite bietet uns die Möglichkeit, so genannte Notebooks zu verwenden, das heißt, interaktive Umgebungen zur einfachen und unmittelbaren Verwendung, die dem Datenanalytiker die Möglichkeit geben, Teile des Codes unabhängig von anderen zu schreiben und auszuführen.
Bevor wir jedoch fortfahren, wollen wir einige theoretische Konzepte vorstellen, die uns helfen, eine gemeinsame Basis für den Diskurs zu schaffen.
Die Konzepte
Das erste Konzept, das man kennen muss, ist das oft als selbstverständlich angesehene Konzept des Datasets, eines Satzes von Samples, von denen jedes durch eine bestimmte Anzahl von Variablen oder Merkmalen gekennzeichnet ist, die das beobachtete Phänomen beschreiben. Der Einfachheit halber können wir uns einen Datensatz wie ein Excel-Sheet vorstellen: In den Zeilen haben wir die Samples, also die einzelnen Beobachtungen des Phänomens, während in den Spalten die Features, die Werte stehen, die jeden der Aspekte des Prozesses charakterisieren. Um auf das Beispiel der intelligenten Fertigung zurückzukommen: Die Zeilen repräsentieren die Bedingungen der Produktionskette zu einem bestimmten Zeitpunkt, während in den Spalten die Messwerte der Sensoren dargestellt werden.
Bei Scikit-Learn spielt das Konzept der Labels oder der Klasse eine Rolle. Das Vorhandensein oder Fehlen von Labels ermöglicht es, zwischen überwachten und nicht überwachten Algorithmen zu unterscheiden. Der Unterschied ist, zumindest im Prinzip, ganz einfach: Überwachte Algorithmen benötigen von vornherein Kenntnisse über die Klasse jedes Samples im Datensatz, während bei unüberwachten Algorithmen dies offensichtlich nicht erforderlich ist. In der Praxis ist es für die Verwendung eines überwachten Algorithmus notwendig, dass ein „Experte“ die Zugehörigkeitsklasse jedes Samples kennt. Bei einem intelligenten Fertigungsprozess könnte dieser Experte feststellen, ob der Datensatz der Messwerte zu einem bestimmten Augenblick eine anomale Situation darstellt oder nicht, und somit die einzelnen Samples eine von zwei möglichen Klassen zuordnen. Für unüberwachte Algorithmen ist dies natürlich nicht notwendig.
Weiterhin muss unterschieden werden zwischen Prozessen mit unabhängigen und identisch verteilten Daten (IID) und mit Daten in einer zeitlichen Reihenfolge. In diesem Fall hängt der Unterschied mit der Art des beobachteten Phänomens zusammen: Die Samples eines IID-Prozesses sind voneinander unabhängig, während in der Zeitreihe jedes Sample von einer linearen oder nichtlinearen Kombination der Werte abhängt, die der Prozess zu vorhergehenden Zeitpunkten angenommen hat.
Lassen Sie uns zur Sache kommen!
Nachdem wir die notwendigen theoretischen und praktischen Begriffe kennengelernt haben, gehen wir zur Praxis über. Wir verwenden einen geeigneten Datensatz zur Beschreibung unseres Beispiels. Dieser Datensatz ist SECOM, ein Akronym, das für SEmiCOnductor Manufacturing steht, ein Datensatz, der die Werte enthält, die von einer Reihe von Sensoren während der Überwachung eines Halbleiterherstellungsprozesses gelesen werden. In dem Datensatz, der von verschiedenen Quellen (wie Kaggle) heruntergeladen werden kann, sind 590 Variablen enthalten, von denen jede repräsentativ für den Messwert eines Sensors zu einem bestimmten Zeitpunkt ist. Der Datensatz enthält auch Labels, die die Fehler und Anomalien vom korrekten Funktionieren des Systems unterscheiden.
Sobald der Datensatz heruntergeladen ist, installieren wir die oben genannten Bibliotheken. Geben Sie dazu in die Befehlszeile ein:
$ pip install numpy pandas scikit-learn matplotlib seaborn jupyter
Schauen wir uns an, wie man eine einfache Pipeline für die Datenanalyse einrichtet.
Das erste Notebook
Der erste Schritt besteht darin, ein neues Notebook einzurichten. Von der Kommandozeile aus starten wir Jupyter mit der folgenden Anweisung:
$ jupyter-notebook
Es öffnet sich ein Bildschirm wie in Bild 1.

Wir erstellen ein Notebook mit New > Python 3. In unserem Browser wird ein Tab mit dem neu erstellten Notebook geöffnet. Machen wir uns mit der Benutzeroberfläche in Bild 2 vertraut, die (sehr vage) einer interaktiven Befehlszeile ähnelt, mit einem Kopfmenü und mehreren Optionen.
Das erste, was auffällt, ist die so genannte Zelle als eines der Teile, in die das Notebook unterteilt ist. Die Ausführung einer einzelnen Zelle wird von der Schaltfläche Run initiiert und ist recht unabhängig von der Ausführung anderer (beachten Sie, dass das Konzept des variablen Bereichs immer gilt).
Mit den drei Schaltflächen rechts neben Run können Sie den Kernel stoppen, neu starten und zurücksetzen. Dies ist die Instanz, die Jupyter unserem Notebook zuordnet. Ein Neustart der Instanz kann insbesondere dann erforderlich sein, wenn man die mit dem Skript verbundenen lokalen und globalen Variablen zurückzusetzen möchte. Dies ist äußerst nützlich, wenn Sie mit neuen Methoden und Bibliotheken experimentieren.
Eine weitere interessante Option ist die Auswahl des Zellentyps, bei der Sie zwischen Code (Python-Code), Markdown (nützlich, um Kommentare und Beschreibungen in dem Format einzufügen, das zum Beispiel für GitHub READMEs verwendet wird), Raw NBContent (reinen Text einzufügen) und Heading (zum Einfügen von Titeln) wählen können.
Daten importieren und anzeigen
Sobald wir mit der Schnittstelle vertraut sind, können wir mit der Implementierung unseres Skripts fortfahren. Wir importieren zunächst die verwendeten Bibliotheken und Module:
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
%matplotlib inline
import seaborn as sns
from ipywidgets import interact
from sklearn.ensemble import RandomForestClassifier
from sklearn.impute import SimpleImputer
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler
from sklearn.metrics import confusion_matrix, accuracy_score
from sklearn.utils import resample
Dabei ist die Anweisung %matplotlib inline hervorzuheben, die nützlich ist, um die von der Matplotlib erzeugten Diagramme korrekt anzuzeigen.
Dann lesen wir die Daten aus dem SECOM-Datensatz mit der Funktion read_csv von Pandas. Eine kurze Anmerkung: In diesem Beispiel ist der relative Pfad zu der Datei der Einfachheit halber fest angegeben, es ist aber in der Praxis ratsam, das Python-Paket os zu verwenden, damit unser Programm diesen Pfad immer frisch ermitteln kann.
data = pd.read_csv('data/secom.csv')
Mit dieser Anweisung lesen wir die in der Datei secom.csv enthaltenen Daten und ordnen sie in ein Dataframe namens data an. In Bild 3 sind die ersten fünf Zeilen des Datenrahmens (plus head()-Anweisung) dargestellt:
data.head()
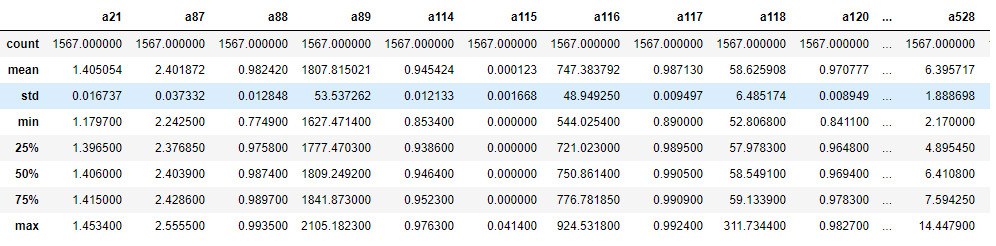
Die Visualisierung der ersten Zeilen des Datenrahmens kann nützlich sein, um einen ersten Überblick über die zu analysierenden Daten zu erhalten: Hier bemerken wir zum Beispiel sofort, dass einige Werte gleich „?“ (vermutliche Null-Werte) vorhanden sind. Darüber hinaus ist es offensichtlich, dass sich die Werte in recht verschiedenen Größenordnungen bewegen, ein Faktor, den wir im Auge behalten müssen. Wir können auch die Funktion describe() verwenden, um einen schnellen Überblick über die statistischen Merkmale der einzelnen Variablen zu erhalten (Bild 4):
data.describe()

Die statistische Analyse kann prinzipiell eine Nicht-Normalität (Daten mit nicht-parametrischer Verteilung) oder das Vorhandensein von Anomalien hervorheben. Um ein Beispiel zu nennen: Wir stellen fest, dass die Standardabweichung der Variablen a116 und a118 verhältnismäßig hoch ist, so dass man erwarten kann, dass diese Daten eine große Bedeutung in der Analyse haben, andererseits haben Variablen wie a114 eine so geringe Varianz, so dass man sie als nicht besonders beachtenswert verwerfen kann.
Sobald das Thema Laden und Anzeigen von Dataframes abgeschlossen ist, können wir zu einem grundlegenden Teil der Pipeline übergehen: dem Preprocessing.
Data Preprocessing
In einem ersten Schritt zeigen wir die Anzahl der mit jeder Klasse verknüpften Samples an. Dazu verwenden wir die Funktion value_counts() in der Spalte classvalue, die die den einzelnen Samples zugeordneten Bezeichnungen enthält.
data['classvalue'].value_counts()
Wir können sehen, dass 1463 Samples in einer „normalen“ Betriebssituation (Klasse -1) und 104 Samples in einer Fehlersituation (Klasse 1) gesammelt wurden. Der Datensatz ist daher stark unausgewogen, und es sollten Vorkehrungen getroffen werden, um die Verteilung der Samples auf die verschiedenen Klassen „einheitlicher“ zu gestalten. Dies hängt mit der intrinsischen Funktionsweise von Algorithmen des maschinellen Lernens zusammen, die auf Grundlage der ihnen zur Verfügung stehenden Daten lernen. In diesem speziellen Fall lernt der Algorithmus, eine Situation als Standardverhalten zu charakterisieren, ist sich aber bei der Charakterisierung anomaler Situationen unsicher. Die Unausgewogenheit wird noch deutlicher, wenn man ein Streudiagramme verwendet (siehe Bild 5):
sns.scatterplot(data.index, data['classvalue'], alpha=0.2)
plt.show()

Behalten wir dieses Ungleichgewicht im Hinterkopf (wir werden später darauf zurückkommen) und „trennen“ nun die Labels von den Daten:
labels = data['classvalue']
data.drop('classvalue', axis=1, inplace=True)
Beachten Sie die Verwendung des Achsenparameters axis in der Drop-Funktion, mit dem man festlegen kann, dass die Funktion mit den Spalten des Datenrahmens arbeiten muss (standardmäßig arbeiten Pandas-Funktionen mit Zeilen).
Eine weitere Information, die aus der Datensatzanalyse gezogen werden kann, ist, dass in dieser speziellen Version der SECOM-Datei viele Spalten unterschiedliche Datentypen enthalten (Strings und numerische Werte). Deshalb ist Pandas nicht in der Lage, den Datentyp eindeutig zu bestimmen und überlässt die Definition dieser Daten dem Benutzer („?“). Um alle Daten in ein numerisches Format zu bringen, werden deshalb drei Pandas-Funktionen genutzt.
Die erste verwendete Funktion ist replace(), mit der wir alle Fragezeichen durch den konstanten Wert numpy.nan ersetzen können, ein Platzhalter, der zur Behandlung von Nullwerten in Numpy-Arrays verwendet wird.
data = data.replace('?', np.nan, regex=False)
Die Syntax der Funktion ist selbsterklärend: Der erste Parameter ist der zu ersetzende Wert, der zweite ist der ersetzende Wert und der dritte ist ein Flag, das angibt, ob der erste Parameter ein regulärer Ausdruck ist oder nicht. Wir könnten auch eine alternative Syntax verwenden, indem wir den inplace-Parameter auf True setzen:
data.replace('?', np.nan, regex=False, inplace=True)
Die beiden anderen Funktionen zur Lösung der oben genannten Probleme sind apply() beziehungsweise to_numeric(). Mit der ersten können Sie eine bestimmte Funktion auf alle Spalten (oder Zeilen) eines Datenrahmens anzuwenden, während die zweite eine einzelne Spalte in numerische Werte umwandelt. Durch ihre Kombination erhalten wir ein eindeutiges Datenformat und entfernen alle für Numpy und Scikit-Learn nicht nachvollziehbaren Werte.
data.apply(pd.to_numeric)
Lassen Sie uns nun bewerten, welche der im Datensatz enthaltenen Funktionen nützlich sind. Normalerweise werden in der Praxis (mehr oder weniger komplexe) Feature-Selection-Techniken verwendet, um Redundanzen und den Umfang des zu behandelnden Problems zu reduzieren. Wegen der offensichtlichen Vorteile in Bezug auf Verarbeitungszeit und die Leistung des Algorithmus werden wir in unserem Fall eine weniger komplexe Technik benutzen, die auf die Eliminierung von Merkmalen mit geringer (und daher, wie oben erwähnt, nicht sehr signifikanter) Varianz baut. Wir erstellen daher ein interaktives Steuerelement, das es uns ermöglicht, die Verteilung der Daten für jedes Merkmal in Form eines Histogramms anzuzeigen:
@interact(col=(0, len(df.columns) - 1)
def show_hist(col=1):
data['a' + str(col)].value_counts().hist(grid=False, figsize=(8, 6))
Die Interaktivität wird durch den Decorator @interact gewährleistet, dessen Referenzwert (col) zwischen 0 und der Anzahl der im Datensatz vorhandenen Merkmale variiert. Wenn wir die angezeigten Daten über das Widget untersuchen, werden wir feststellen, wie viele Merkmale nur einen einzigen Wert annehmen und damit bei der Analyse keine Rolle spielen. Wir können sie deshalb eliminieren:
single_val_cols = data.columns[len(data)/data.nunique() < 2]
secom = data.drop(single_val_cols, axis=1)
Natürlich gibt es relevantere und raffiniertere Techniken zur Feature Selection, die beispielsweise auf statistischen Parametern basieren. Einen vollständigen Überblick bietet die Dokumentation bei Scikit-Learn [5].
Der letzte Schritt besteht darin, sich mit Nullwerten zu befassen (die wir zuvor durch np.nan ersetzt haben). Wir untersuchen Datensatz, um zu sehen, wie viele es davon gibt. Dazu verwenden wir eine Heatmap wie in Bild 6, wobei die hellen Stellen die Nullwerte darstellen.

sns.heatmap(secom.isnull(), cbar=False)
Es ist offensichtlich, dass viele Samples einen hohen Prozentsatz von Nullwerten aufweisen, so das sie nicht berücksichtigt werden sollten, um einen Bias- oder Verzerrungseffekt zu vermeiden.
na_cols = [col for col in secom.columns if secom[col].isnull().sum() / len(secom) > 0.4]
secom_clean = secom.drop(na_cols, axis=1)
secom_clean.head()
Dank der obigen Anweisungen erstellen wir eine „list comprehension“, um alle Features mit mehr als 40% Nullwerten zu isolieren und sie später aus dem Datensatz zu entfernen.
Nur die Features mit weniger als 40% Nullwerten müssen noch behandelt werden. Dazu verwenden wir unser erstes Scikit-Learn-Objekt SimpleImputer, das allen NaNs auf der Grundlage einer benutzerdefinierten Strategie Werte zuweist. Hier kommt die Mittelwertstrategie (mean) zum Einsatz. Jedem NaN wird der vom Feature angenommene Mittelwert zugeordnet.
imputer = SimpleImputer(strategy='mean')
secom_imputed = pd.DataFrame(imputer.fit_transform(secom_clean))
secom_imputed.columns = secom_clean.columns
Zur Übung können wir eine weitere Heatmap erstellen, die – vorhersehbar - eine einheitlich dunkle Farbe annehmen wird, um zu bestätigen, dass es keinen Nullwert im Datensatz mehr gibt. Damit kommen wir zur eigentlichen Datenverarbeitung.
Datenverarbeitung
Wir teilen unseren Datensatz in die beiden Untergruppen Training und Test auf. Diese Unterteilung ist notwendig, um das Phänomen der Überanpassung abzuschwächen, das den Algorithmus „zu sehr an den Daten haften“ lässt (weitere Informationen unter [6]). Dadurch wird sichergestellt, dass das gefundene Modell auch auf andere Fälle als den, an dem es trainiert wurde, anwendbar ist. Dazu verwenden wir die Funktion train_test_split:
X_train, X_test, y_train, y_test = train_test_split(secom_imputed, labels, test_size=0.3)
Mit dem Parameter test_size können wir den Prozentsatz der für den Test reservierten Daten angeben. Die Standardwerte für diesen Parameter liegen normalerweise zwischen 0,2 und 0,3.
Eine weitere wichtige Aufgabe ist die Normalisierung der Daten. Wir haben bereits bemerkt, dass bei einigen Features Werte in viel größeren Bereichen annehmen als andere, was ihnen auch mehr Gewicht in der Analyse verleihen würde. Eine Normalisierung erlaubt es, alle Features mit einem einheitlichen Wertebereich zu versehen, so dass es keine Ungleichgewichte aufgrund von anfänglichen Offsets gibt. Dazu verwenden wir einen StandardScaler:
scaler = StandardScaler()
X_train = pd.DataFrame(scaler.fit_transform(X_train), index=X_train.index, columns=X_train.columns)
X_test = pd.DataFrame(scaler.fit_transform(X_test), index=X_test.index, columns=X_test.columns)
Hier macht sich die von Scikit-Learn angebotene gemeinsame Schnittstelle nützlich: Sowohl Scaler als auch Imputer verwenden die Methode fit_transform, um Daten zu verarbeiten, was in komplexen Pipelines das Schreiben von Code und das Verständnis der Bibliothek stark vereinfacht.
Wir sind nun endlich bereit, die Daten zu klassifizieren. Wir verwenden einen Random Forest [7], um nach dem Training ein Modell zu erhalten, das in der Lage ist, zwischen normalen und anormalen Situationen zu unterscheiden. Wir überprüfen die Leistungsfähigkeit des ausgearbeiteten Modells auf zwei Arten, zum einen durch den Accuracy Score, dem Prozentsatz der Samples im Testsatz, die durch den Algorithmus korrekt klassifiziert wurden, zum anderen durch die Wahrheits- oder Konfusionsmatrix [8], die es erlaubt, die Anzahl der falsch positiven und falsch negativen Ergebnisse zu unterscheiden.
Zuerst erstellen wir den Klassifikator:
clf = RandomForestClassifier(n_estimators=500, max_depth=4)
Wir verwenden einen Random Forest mit 500 Estimators (die Anzahl der Bäume im Wald) mit einer maximalen Tiefe von vier Ebenen. Jetzt können wir unser Modell anhand der Trainingsdaten trainieren:
clf.fit(X_train, y_train)
Sobald das Training abgeschlossen ist, lässt sich das trainierte Modell zur Klassifizierung der Testsamples verwenden:
y_pred = clf.predict(X_test)
Wir verfügen dann für jedes Sample der Testmenge über zwei Labels: Das erste (zu y_test gehörende) Label repräsentiert die „Wahrheit“, während das zweite (zu y_pred gehörende) Label den vom Algorithmus vorhergesagten Wert darstellt. Wenn wir sie vergleichen, erhalten wir sowohl die Accuracy als auch die Confusion_matrix.
accuracy = accuracy_score(y_test, y_pred))
cf = confusion_matrix(y_test, y_pred))
Im Beispieldurchlauf haben wir die folgenden Ergebnisse erhalten:
Die Modellgenauigkeit des Testsatzes beträgt: 0.9341825902335457
Die Konfusionsmatrix des Modells ist:
[[440 0]
[ 31 0]]
Die Genauigkeit von etwa 93% ist sehr gut, so dass das Modell in Ordnung zu sein scheint. Wir stellen jedoch fest, dass das Modell zwar bei der Klassifizierung der Samples der tonangebenden Klasse sehr genau ist, aber ebenso ungenau bei der Klassifizierung der Samples, die der „Minderheitenklasse“ angehören. Es ist offensichtlich eine Verzerrung vorhanden.
Wir sollten und können eine Strategie finden, um diese Situation zu verbessern. Dazu setzen wir ein Upsampling der Daten fort, die der Minderheitenklasse angehören, um den Datensatz zumindest teilweise auszugleichen. Dazu eignet sich die Resample-Funktion von Pandas.
normals = data[data['classvalue'] == -1]
anomalies = data[data['classvalue'] == 1]
anomalies_upsampled = resample(anomalies, replace=True, n_samples=len(normals))
Wir erhöhen den Umfang des Datensatzes, um die Anzahl der normalen Samples an die Anzahl der anomalen Samples anzupassen. Daher müssen sowohl X als auch Y neu definiert werden:
upsampled = pd.concat([normals, anomalies_upsampled])
X_upsampled = upsampled.drop('classvalue', axis=1)
y_upsampled = upsampled['classvalue']
Durch erneutes Durchführen des Trainings (und natürlich durch Wiederholung der Split- und der Normalisierungsprozedur) erhalten wir in unserem Beispieldurchlauf die folgenden Ergebnisse:
Die Modellgenauigkeit des Testsatzes beträgt: 0,8631921824104235
Die Konfusionsmatrix des Modells ist:
[[276 41]
[ 43 254]]
Wir stellen sofort fest, dass die Genauigkeit des Modells abgenommen hat, was vermutlich auf die nun größere Heterogenität des Datensatzes zurückzuführen ist. Doch wenn man die Konfusionsmatrix betrachtet, fällt auf, dass das Modell seine Verallgemeinerungsfähigkeiten tatsächlich verbessert hat und es gelingt, auch Samples, die zu anomalen Situationen gehören, korrekt zu klassifizieren.
Schlussfolgerungen und Hinweise
In diesem Artikel haben wir eine Pipeline für die Analyse von Daten aus realen Prozessen in Python kennengelernt. Es ist jedoch klar, dass jedes der behandelten Themen in der Tat äußerst vielfältig ist und theoretische wie praktische Kenntnisse unerlässlich sind, wenn man sich ernsthaft mit der Datenanalyse beschäftigen möchte. Wir haben auch gelernt, dass man nicht beim ersten erzielten Ergebnis stehen bleiben darf, auch nicht in so komplexen Situationen wie der behandelten: Es ist vielmehr notwendig, die erzielten Ergebnisse aus verschiedenen Blickwinkeln zu interpretieren, um den Unterschied zwischen einem arbeitsfähigen und einem mehr oder weniger offensichtlich von Verzerrungen betroffenen Modell zu entdecken. Die Botschaft, die es mit nach Hause zu nehmen gilt, lautet daher: Datenanalyse ist keine mechanische Disziplin, sondern setzt eine kritische, eingehende und vielfältige Analyse des beobachteten Phänomens voraus, die sich an theoretischen Vorstellungen und praktischen Fertigkeiten orientiert. Abschließend finden Sie in den Weblinks einige Hinweise, durch die Sie einige der im Artikel angesprochenen Aspekte vertiefen können, sowie den Link zum GitLab-Repository, wo Sie den für den Artikel geschriebenen Code einsehen können.
(200505-02)
Wir haben diesen Beitrag der Zeitschrift/Online-Plattform Elettronica Open Source entnommen und mit freundlicher Genehmigung des Verlags übersetzt (https://it.emcelettronica.com).
Wollen Sie weitere Elektor-Artikel lesen? Jetzt Elektor-Mitglied werden und nichts verpassen!


Diskussion (3 Kommentare)